发布时间:2022/04/22 点击数:
发布时间:2022/04/22 点击数:对单个细胞的RNA进行大规模测序可以揭示不同细胞类型和细胞状态下的基因、异构体以及等位基因的表达模式差异。具有完整转录本覆盖率的基于孔板的单细胞RNA测序方法(SMART-Seq方法)通常在灵敏度方面表现出色,但成本更高且更耗时。瑞典卡罗林斯卡学院的Rickard团队,于2020年5月6日,在smart-seq2的基础上,通过改进开发了smart-seq3技术(Single-cell RNA counting at allele and isoform resolution using Smart-seq3),该技术就是低成本的单细胞全长转录组的解决方案,并将该技术发表在Nature Biotechnology 。
时隔一年,于2021年7月,该团队继续获得研究新突破,在smart-seq3的基础上,对该流程进行mini化和简化,开发smart-seq3xpress方法以降低成本并提高样品处理量。
cDNA合成反应体系
该团队首先探究了反应体系的大小对文库复杂程度的影响,结果如下:

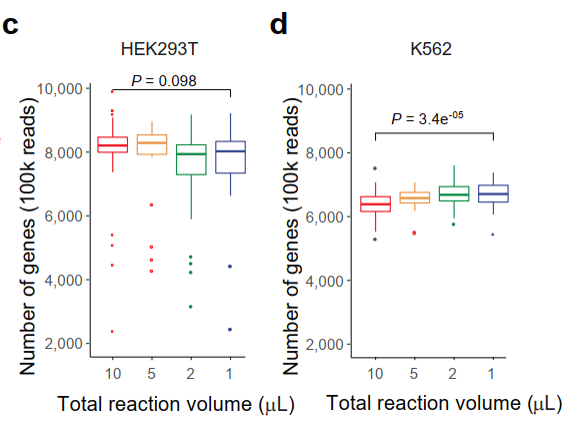
图1
(a)在384孔PCR板的孔中进行纳升级cDNA合成反应示意图(带有3µL overlay)
(b)具体的lysis、RT mixbuffer、PCR mixbuffer在不同反应体系中的具体用量说明。
(c)在HEK293T细胞和K562细胞
(d)相同测序深度下(100K reads),不同反应体系对应检测到的基因数目。P值代表10ul和1ul反应体系之间的t测验结果。
随后,作者探究了低反应体系(Low Volume)中使用不同的overlay(作用:防蒸发)对文库质量的影响,结果如下:

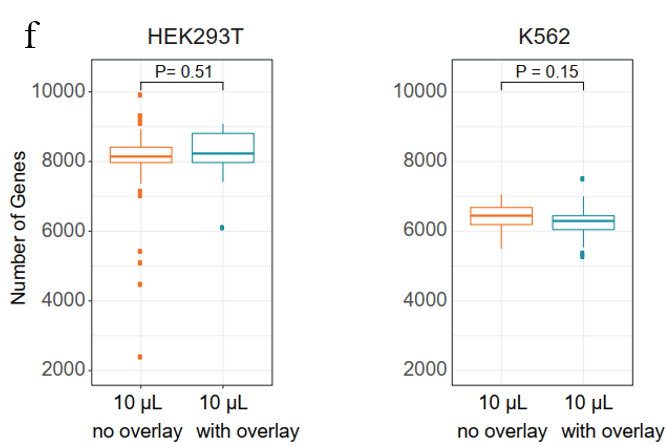
图2
(e)测试了不同材质的overlay对文库复杂程度的影响,针对小反应体系,不使用overlay会造成文库库容量的严重丢失;
(f)测试了overlay的使用并不会对文库质量产生干扰。
以上数据表明,在可以有效预防蒸发的前提下缩小反应体系(10ul缩小至1ul)并不影响文库数据质量。
标记反应
基于孔板的单细胞转录组测序方法,由于每个细胞均需要单独进行标记(Tagmentation),导致成本增加,方法之一是通过减少Tn5试剂(标记反应所用到的酶)的使用量来降低成本,因此该团队对Tn5试剂的用量以及cDNA用量与文库的复杂性之间的相关性进行探究。实验结果如下:
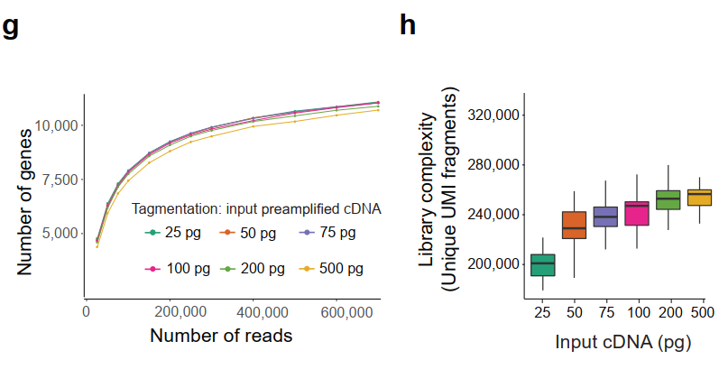
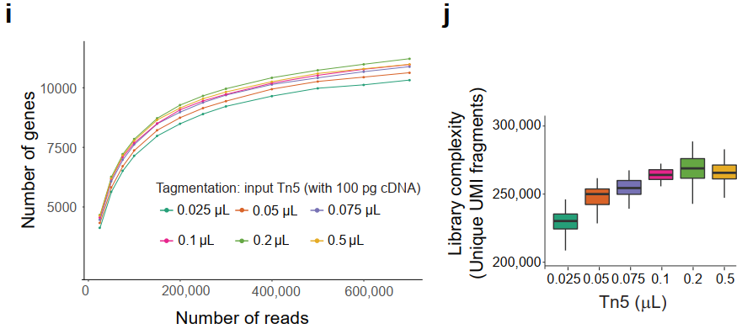
图3
(g)input的cDNA容量大小对文库复杂程度的影响(基因数目);
(h)相同测序深度(reads)下,同一个cDNA产物不同使用量可获得的UMI片段数量 ;
(i)Tn5试剂的用量对文库复杂程度的影响(基因数目);
(j)相同测序深度(reads)下,不同Tn5试剂用量可获得的UMI片段数量
这些结果表明,标记反应在cDNA输入和Tn5使用量的较大变化中是稳健的,因此可通过减少Tn5用量来节约成本,并且不会影响文库复杂性。
简化流程
在此理论依据基础之上,作者认为可以排除一些耗时且昂贵的实验步骤,包括过度的cDNA预扩增、cDNA纯化步骤(Clean-up)、QC、浓度检测及均一化。对比Smart-seq3,Smart-seq3xpress简化流程请见下图:

图4 Smart-seq3和Smartseq3xpress工作流程的示意图
该团队对cDNA预扩增循环数量以及纯化流程对文库复杂程度的影响进行了探究,实验结果如下:
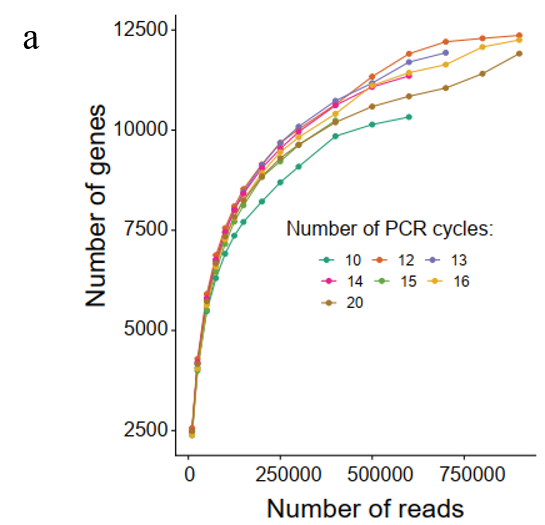
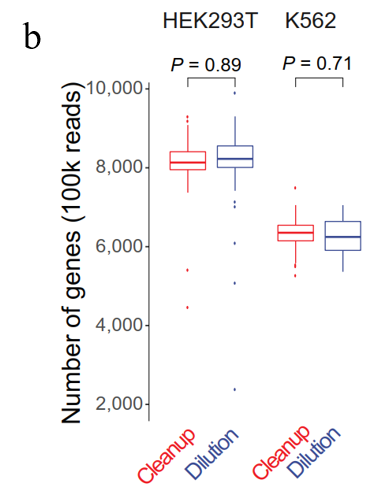
图5
(a)一系列cDNA扩增循环数对应可检测到的基因数目(low-volume RT,1ul;HEK293FT cells;n≥8);
(b)研究了在较低体积中是否可以通过稀释获得的cDNA来替代耗时的cDNA纯化步骤。在相同的测序深度下,通过cDNA稀释和纯化产生的单细胞文库无可检测的差异(t检验,分别为p=0.89和p=0.71)
以上数据表明,减少cDNA扩增循环数以及酶增反应后获得的cDNA产物无需纯化即可直接用于后续的标记反应。
5’ UMI偏好性
Smart-seq3方法获得的库严重偏向于包含UMI的5 '读本,而牺牲了内部(internal)读本,而内部读本对于获得完整的转录组覆盖率至关重要。该研究团队测试了6种不同的商用聚合酶,对后续标记反应的兼容性。测试结果如下:

图6 使用不同的聚合酶进行cDNA扩增,最终测序读取到的5‘ UMI的百分比
使用SeqAmp和PlatinumII扩增的cDNA进行标记反应,后续得到的检测数据显示显著降低了5'UMI读数的比例,降低文库构建的偏好性。
Conclusion
2021年7月,瑞典卡罗林斯卡学院的Rickard团队,研发出Smart-seq3xpress,简化耗时且昂贵的建库步骤,缩小反应体系,降低成本的同时提高样本处理量。
实验数据表明,获得cDNA过程中,在防止蒸发的前提下缩小反应体系至原来的1/10(10ul-1ul),减少cDNA扩增的PCR循环数,扩增时间,去除耗时的cDNA纯化步骤,直接稀释进行后续的标记反应均不会影响后续的文库质量。实验发现,标记反应在cDNA输入和Tn5使用量的较大变化中是稳健的,因此可通过减少Tn5用量来节约成本,并且不会影响文库复杂性。此外,使用特定的聚合酶如SeqAmp和PlatinumII,扩增的cDNA进行标记反应,可降低5'UMI读数的比例,得到更优化的测序数据。
经
过一系列简化实验步骤和缩小昂贵试剂使用量,在不影响文库质量以及测序数据的准确性的前提下,相比较成熟的Smart-seq2技术,在Smart-seq3将成本降低十几倍的基础上,Smart-seq3xpress可以实现成本再次下降3-4倍(图7)
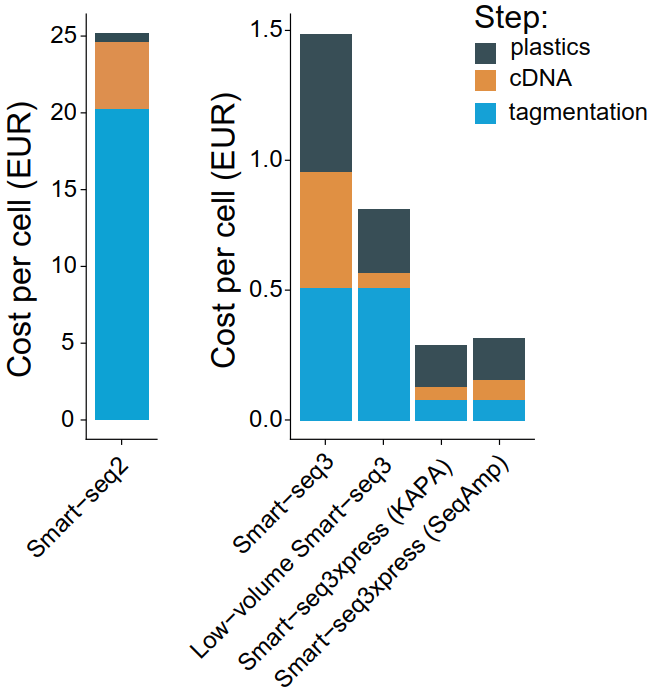
图7 不同建库方法对应的成本
NOTE!!!
实验中低至1ul的反应体系,其中部分组分已达到纳升级,要想实现这么低体积的准确分液同时还要保证高通量,我们的I-DOT-非接触式微量分液系统就要派上用场啦!
I-DOT 非接触式微量分液系统